Martini(粗視化)力場を用いた計算(チュートリアル)
Martiniのあれこれ
タンパク質のシミュレーション
- 全原子モデルから粗視化モデルへ
- 適切なMartiniトポロジーファイルの生成
- タンパク質の水和
1と2は、Martini研が配布しているmartinize.py(python2.6,2.7しか、動作を保証していない)を使う。前提として、Gromacsのコマンドラインは使えることだそうです。
チュートリアル
Martiniトポロジーファイルを作るために、まずはタンパク質を粗視化する。
martinize.pyを使うが、分からないことが有ればヘルプを見ることが出来る。
$python martinize.py -h
早速、粗視化モデルを作る。Martiniの粗視化モデルでは、DSSPの二次構造情報ファイルが必要となるのであらかじめ用意しておく。このブログでDSSPと検索すれば出てきます。データベースを全てダウンロードしようとすると、容量が大きくなり過ぎるので注意してください。
$python martinize.py -f 1AKI.pdb -o system-vaccum.top -x 1ALK-CG.pdb -ss dssp/1aki.dssp -p backbone -ff martini22
上記のコマンドを実行すると、system-vaccum.top,1AKI-CG.pdb,Protein_A.itpが生成される。system-vaccum.topの一行目を変える。martini_v2.2.itpはここに入っています。
#include "martini_v2.2.itp"
#include "Protein_A.itp"
[ system ]
; name
Martini system from 1AKI.pdb
[ molecules ]
; name number
Protein_A 1
Energy最小化をする。
minimization-vaccum.mdp
integrator = steep dt = 0.02 nsteps = 10 nstxout = 0 nstvout = 0 nstlog = 100 nstxtcout = 100 xtc-precision = 10 rlist = 1.4 coulombtype = shift rcoulomb = 1.2 epsilon_r = 15 vdw-type = shift rvdw-switch = 0.9 rvdw = 1.2 tcoupl = v-rescale tc-grps = Protein tau-t = 1.0 ref-t = 300
以上のファイルを使って、
$grompp -f minimization-vaccum.mdp -p system-vaccum.top -c 1ALK-CG.pdb -o minimization-vaccum.tpr $mdrun -deffnm minimization-vaccum -v
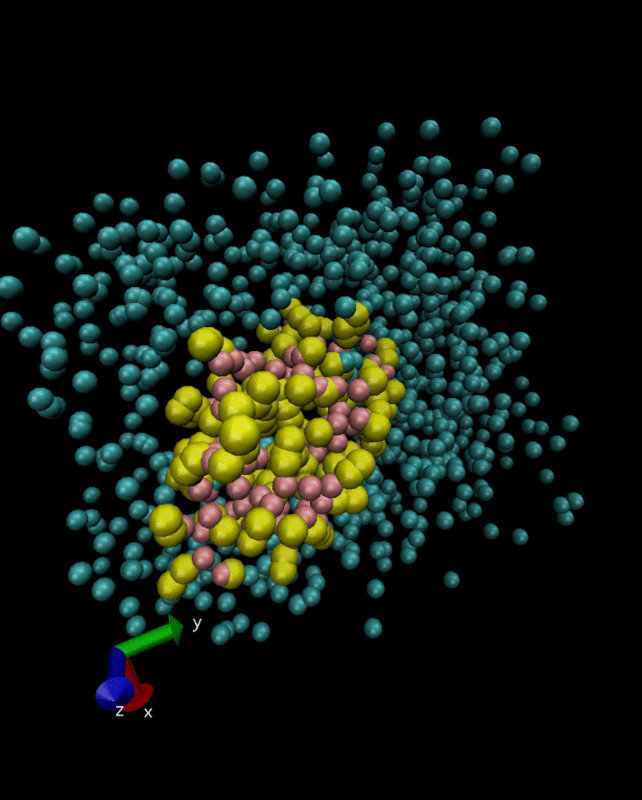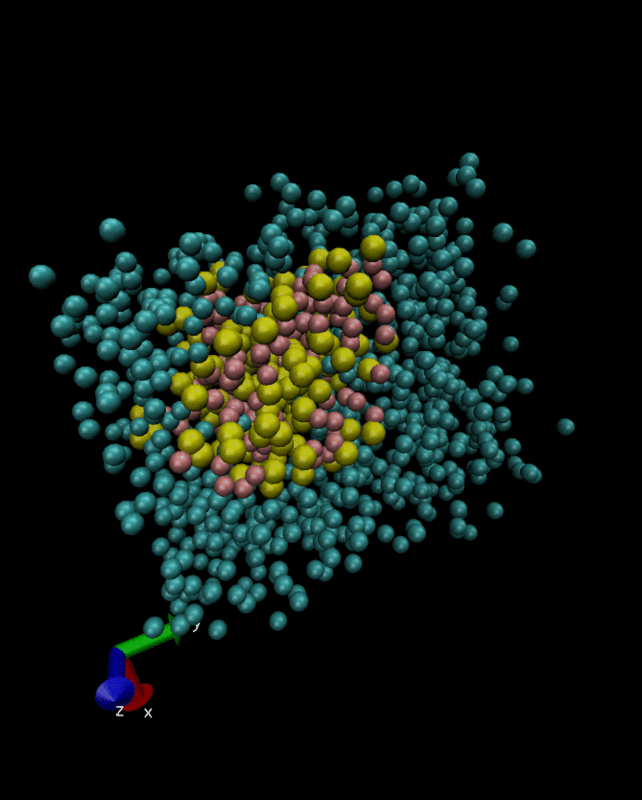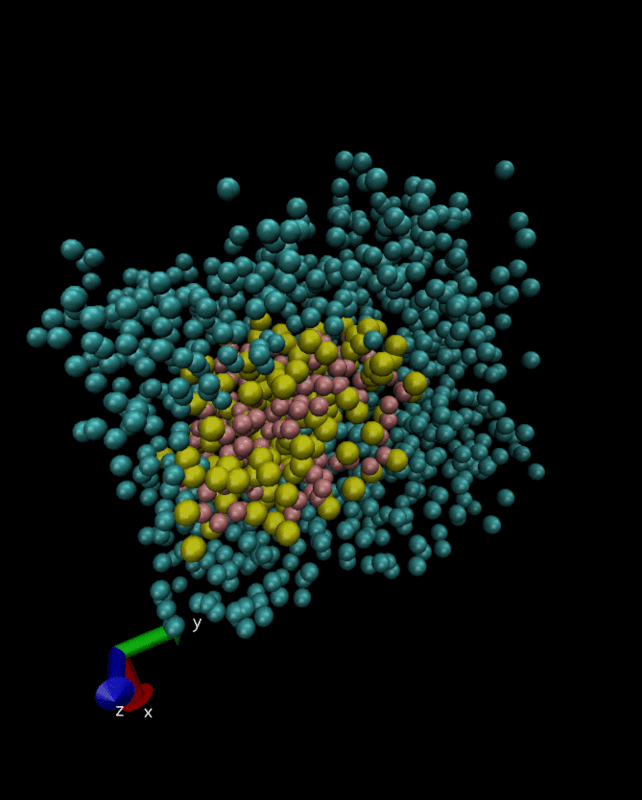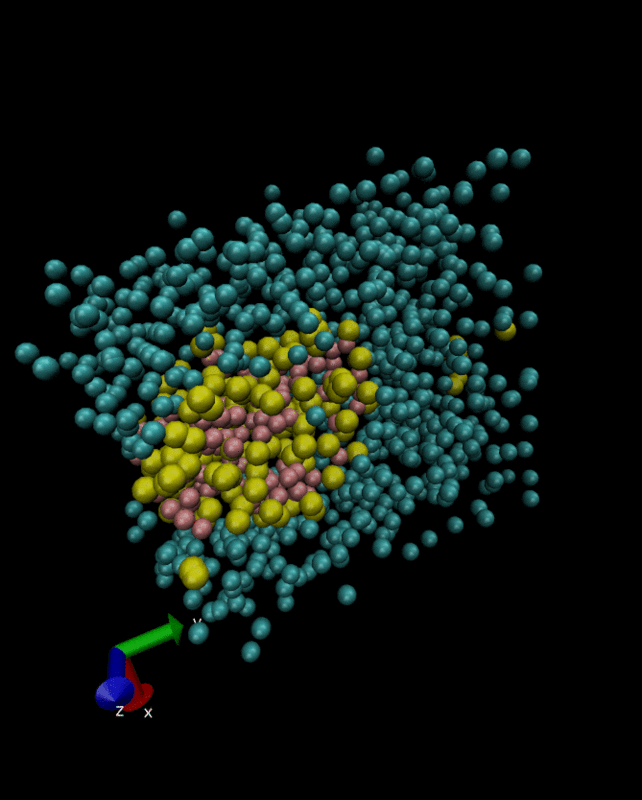
- 次に、水和させるが必要な水のモデルはここからダウンロードする。
genbox -cp minimization-vaccum.gro -cs water.gro -vdwd 0.21 -o system-solvated.gro
また、Energy最小化する。
minimization.mdp
integrator = steep dt = 0.02 nsteps = 500 nstxout = 0 nstvout = 0 nstlog = 100 nstxtcout = 100 xtc-precision = 10 rlist = 1.4 coulombtype = shift rcoulomb = 1.2 epsilon_r = 15 vdw-type = shift rvdw-switch = 0.9 rvdw = 1.2 tcoupl = v-rescale tc-grps = Protein W tau-t = 1.0 1.0 ref-t = 300 300
上記のファイルを使って、
$grompp -f minimization.mdp -c system-solvated.gro -p system.top -o minimization.tpr $mdrun -deffnm minimization -v
systemo.topは以下のようにした。このファイルにおける水の数(W)はsystem-solvated.groの水の数に合わせた。
#include "martini_v2.2.itp"
#include "Protein_A.itp"
[ system ]
; name
Martini system from 1AKI.pdb
[ molecules ]
; name number
Protein_A 1
W 722
系を平衡化する。
define = -DPOSRES dt = 0.02 nsteps = 25000 nstxout = 0 nstvout = 0 nstlog = 100 nstxtcout = 100 xtc-precision = 10 rlist = 1.4 coulombtype = shift rcoulomb = 1.2 epsilon_r = 15 vdw-type = shift rvdw-switch = 0.9 rvdw = 1.2 tcoupl = v-rescale tc-grps = Protein W tau-t = 1.0 1.0 ref-t = 300 300 Pcoupl = parrinello-rahman Pcoupltype = isotropic tau-p = 12.0 compressibility = 3e-4 ref-p = 1.0 refcoord_scaling = all
上記の内容をもつequilibration.mdpを使って、
$grompp -f equilibration.mdp -c minimization.gro -p system.top -o equilibration.tpr $mdrun -deffnm equilibration -v -rcon 1.25
rconオプションに関してはよくわかっていません。エラーがでたので、指示されている通りに修正しました。拘束オプションとボックスサイズがうまく噛み合ってないらしいです。
本番の計算!
dt = 0.02 nsteps = 1000000 nstxout = 0 nstvout = 0 nstlog = 10000 nstxtcout = 1000 xtc-precision = 10 rlist = 1.4 coulombtype = shift rcoulomb = 1.2 epsilon_r = 15 vdw-type = shift rvdw-switch = 0.9 rvdw = 1.2 tcoupl = v-rescale tc-grps = Protein W tau-t = 1.0 1.0 ref-t = 300 300 Pcoupl = parrinello-rahman Pcoupltype = isotropic tau-p = 12.0 compressibility = 3e-4 ref-p = 1.0 refcoord_scaling = all
上記の内容のdynamic.mdpを使って、
$grompp -f dynamic.mdp -c equilibration.gro -p system.top -o dynamic.tpr $mdrun -deffnm dynamic -v -rcon 1.25
何か変(笑)